Spliter et fusionner un fichier MED grâce à l’API avancée de MEDLoader¶
Objectif¶
Cet exercise présente un cas complet et avancé d’utilisation de l’API avancée de MEDLoader. Le but est de créer un maillage multi-type à partir de rien avec 2 champs :
- un champ aux cellules “CellField”
- un champ aux noeuds “NodeField”
Nous allons ensuite couper ces champs en deux parties (dans le but d’un traitement en parallèle par un code par exemple) et aussi montrer comment re-fusionner deux champs à partir de morceaux disjoints.
Début de l’implémentation¶
Créer un unstructured mesh m0 issu d’un maillage structuré (meshDim=2, spaceDim=2) de 30*30. Chacune des cellules paires du maillage sera simplexisée (i.e. coupée en triangle - méthode MEDCouplingUMesh.simplexize(0))
import MEDLoader as ml
m0 = ml.MEDCouplingCMesh()
arr = ml.DataArrayDouble(31,1) ; arr.iota(0.)
m0.setCoords(arr,arr)
m0 = m0.buildUnstructured()
m00 = m0[::2] # Extract even cells
m00.simplexize(0)
m01 = m0[1::2]
m0 = ml.MEDCouplingUMesh.MergeUMeshes([m00,m01])
m0.getCoords()[:] *= 1/15. # Illustrate how to quickly rescale a mesh
m0.setName("mesh")
Note
Le setName() sur “m0” est obligatoire. Ne pas oublier que dans le contexte MED fichier le nommage correct des maillages est fondamental.
Créer les champs cellField et nodeField au pas de temps identifié à (5,6) et au pas de temps 5.6 s.
# Cell field
cellField = ml.MEDCouplingFieldDouble(ml.ON_CELLS, ml.ONE_TIME)
cellField.setTime(5.6,5,6)
cellField.setMesh(m0)
cellField.setName("CellField")
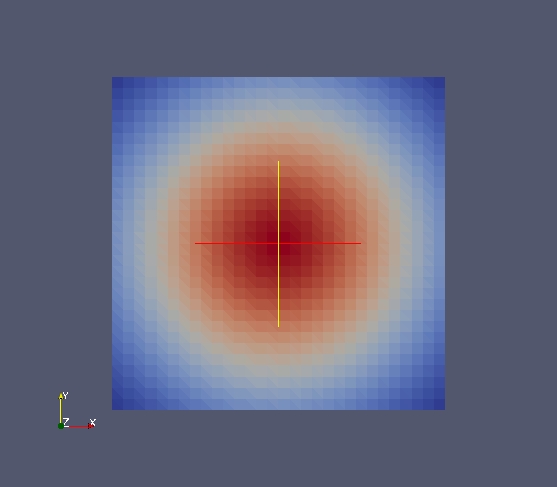
cellField.fillFromAnalytic(1,"exp(-((x-1)*(x-1)+(y-1)*(y-1)))")
cellField.getArray().setInfoOnComponent(0,"powercell [W]")
# Node field
nodeField = ml.MEDCouplingFieldDouble(ml.ON_NODES,ml.ONE_TIME)
nodeField.setTime(5.6,5,6)
nodeField.setMesh(m0)
nodeField.setName("NodeField")
nodeField.fillFromAnalytic(1,"exp(-((x-1)*(x-1)+(y-1)*(y-1)))")
nodeField.getArray().setInfoOnComponent(0,"powernode [W]")
On obtient par exemple pour “CellField” ceci :
Partitionnement de maillage¶
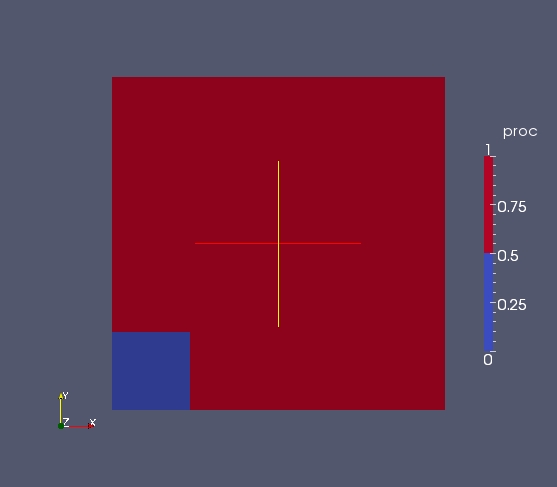
Couper m0 en deux parties distinctes. Les deux parties seront nommées proc0 et proc1. proc0 sera la partie dans la boîte englobante (MEDCouplingUMesh.getCellsInBoundingBox()) [(0.,0.4),(0.,0.4)] à 1e-10 près. proc1 sera le complémentaire (DataArrayInt.buildComplement()).
proc0 = m0.getCellsInBoundingBox([(0.,0.4),(0.,0.4)],1e-10)
proc1 = proc0.buildComplement(m0.getNumberOfCells())
Ecriture dans 2 fichiers MED séparés¶
En partant du partitionnement proc0 et proc1 créer 2 fichiers MED appelés “proc0.med” et “proc1.med” :
nodeField0 = nodeField[proc0] ; cellField0 = cellField[proc0] ; cellField0.setMesh(nodeField0.getMesh())
nodeField1 = nodeField[proc1] ; cellField1 = cellField[proc1] ; cellField1.setMesh(nodeField1.getMesh())
proc0_fname = "proc0.med"
ml.WriteField(proc0_fname, nodeField0, True)
ml.WriteFieldUsingAlreadyWrittenMesh(proc0_fname, cellField0)
proc1_fname = "proc1.med"
ml.WriteField(proc1_fname,nodeField1,True)
ml.WriteFieldUsingAlreadyWrittenMesh(proc1_fname,cellField1)
Lecture et fusion des 2 fichiers MED séparés (non optimal)¶
Partant de “proc0.med” et de “proc1.med” lire leur “CellField” respectif avec l’API basique, agréger les deux et mettre le résultat dans cellField_read :
cellField0_read = ml.ReadFieldCell("proc0.med","mesh",0,"CellField",5,6)
cellField1_read = ml.ReadFieldCell("proc1.med","mesh",0,"CellField",5,6)
cellField_read = ml.MEDCouplingFieldDouble.MergeFields([cellField0_read,cellField1_read])
Note
On peut avoir l’impression que l’information Cell (méthode ReadFieldCell) est répétée de manière abusive (effectivement le champ “CellField” a été créé aux cellules), mais ne pas oublier que dans la norme MED fichier rien n’interdit qu’un champ repose sur des cellules mais aussi simultanément sur des noeuds, ou des points de Gauss ...
Comparer cellField_read et cellField0. Problème, à cause de la contrainte sur la numérotation MED fichier, on a perdu la numérotation initiale. Ou plus exactement il n’y a pas de moyen standard de retrouver la numérotation originale. Donc un MEDCouplingFieldDouble.isEqual() n’est pas suffisant. Utilisons un MEDCouplingFieldDouble.substractInPlaceDM() qui opère pour nous une renumérotation suivant une politique particulière (policy, voir doc html). Pour ce faire une copie profonde (deep copy) de cellField vers cellFieldCpy et opérer sur cette copie un substractInPlaceDM (DM pour “Different Meshes”, contrairement à substract qui ne marche que s’ils partagent le même maillage):
cellFieldCpy = cellField.deepCopy()
cellFieldCpy.substractInPlaceDM(cellField_read,10,1e-12)
cellFieldCpy.getArray().abs()
print cellFieldCpy.getArray().isUniform(0.,1e-12)
Opérons le même travail sur “NodeField” que celui réalisé plus haut sur “CellField”. La différence ici c’est qu’il va y avoir duplication de l’information à la frontière, car les noeuds limites sont partagés des deux côtés :
nodeField0_read = ml.ReadFieldNode("proc0.med","mesh",0,"NodeField",5,6)
nodeField1_read = ml.ReadFieldNode("proc1.med","mesh",0,"NodeField",5,6)
nodeField_read = ml.MEDCouplingFieldDouble.MergeFields([nodeField0_read, nodeField1_read])
Note
Dans cette partie, on a donc relu le maillage une deuxième fois ce qui peut être pénalisant ...
Invoquer MEDCouplingUMesh.mergeNodes() sur nodeField_read pour lui retirer les noeuds dupliqués. Faire une deep copy appelée nodeFieldCpy de nodeField et supprimer encore les doublons :
nodeField_read.mergeNodes(1e-10)
nodeFieldCpy = nodeField.deepCopy()
nodeFieldCpy.mergeNodes(1e-10)
Note
A noter que mergeNodes() possède deux paramètres de précisions (epsilons), le premier, classique, sur la distance absolue entre les noeuds, et l’autre sur la tolérance acceptée sur les valeurs du champ. Si la valeur du champ de deux noeuds à fusionner dépasse ce deuxième epsilon, une exception est levée.
Comparer nodeFieldCpy et nodeField_read toujours en utilisant MEDCouplingFieldDouble.substractInPlaceDM() :
nodeFieldCpy.substractInPlaceDM(nodeField_read,10,1e-12)
print nodeFieldCpy.getArray().isUniform(0.,1e-12)
Lecture et merge des 2 fichiers MED séparés (moins facile, mais plus optimal)¶
Il s’agit ici de faire une méthode plus systématique et potentiellement plus générale de fusion de fichiers. Pour de gros fichiers cette approche est à préférer. Outre la performance, cette approche a l’avantage de pouvoir rajouter des infos.
Avec l’API avancée lire les maillages des deux fichiers “proc0.med” et “proc1.med” et agréger le résultat dans une instance mergeMLMesh de MEDFileUMesh. Traiter tous les niveaux de dimension (même si ici il n’y en a qu’un seul) en utilisant la méthode MEDFileUMesh.getNonEmptyLevels() sur l’instance venant de “proc0.med”.
La solution donnée ci-dessous est la plus générique possible, car elle traite aussi les différents pas de temps et les différents types géométriques :
fileNames = ["proc0.med","proc1.med"]
msML = [ml.MEDFileMesh.New(fname) for fname in fileNames]
fsML = [ml.MEDFileFields.New(fname) for fname in fileNames]
mergeMLMesh = ml.MEDFileUMesh()
mergeMLFields = ml.MEDFileFields()
for lev in msML[0].getNonEmptyLevels():
o2nML = len(msML[0].getNonEmptyLevels())*[None]
cs = [mML.getCoords() for mML in msML]
mergeMLMesh.setCoords(ml.DataArrayDouble.Aggregate(cs))
ms = [mML.getMeshAtLevel(lev) for mML in msML]
m = ml.MEDCouplingUMesh.MergeUMeshes(ms) ; m.setCoords(mergeMLMesh.getCoords())
o2nML[lev] = m.sortCellsInMEDFileFrmt()
mergeMLMesh.setMeshAtLevel(lev,m)
pass
for fieldName in fsML[0].getFieldsNames():
fmts = [fML[fieldName] for fML in fsML]
mergeField = ml.MEDFileFieldMultiTS()
for dt,it,tim in fmts[0].getTimeSteps():
fts = [fmt[dt,it] for fmt in fmts]
arrs = len(fts)*[None]
for typp in fts[0].getTypesOfFieldAvailable():
arr1s = []
if typp == ml.ON_CELLS:
for ft in fts:
for geoTyp,smth in ft.getFieldSplitedByType():
if geoTyp != ml.NORM_ERROR:
smth1 = filter(lambda x:x[0] == ml.ON_CELLS,smth)
arr2s = [ft.getUndergroundDataArray()[elt[1][0]:elt[1][1]] for elt in smth1]
arr1s.append(ml.DataArrayDouble.Aggregate(arr2s))
pass
pass
pass
pass
else:
for ft in fts:
smth = filter(lambda x:x[0] == ml.NORM_ERROR,ft.getFieldSplitedByType())
arr2 = ml.DataArrayDouble.Aggregate([ft.getUndergroundDataArray()[elt[1][0][1][0]:elt[1][0][1][1]] for elt in smth])
arr1s.append(arr2)
pass
pass
arr = ml.DataArrayDouble.Aggregate(arr1s)
if typp == ml.ON_CELLS:
arr.renumberInPlace(o2nML[lev])
mcf = ml.MEDCouplingFieldDouble(typp,ml.ONE_TIME) ; mcf.setName(fieldName) ; mcf.setTime(tim,dt,it) ; mcf.setArray(arr)
mcf.setMesh(mergeMLMesh.getMeshAtLevel(lev)) ; mcf.checkConsistencyLight()
mergeField.appendFieldNoProfileSBT(mcf)
pass
pass
mergeMLFields.pushField(mergeField)
pass
mergeMLMesh.write("merge.med",2)
mergeMLFields.write("merge.med",0)