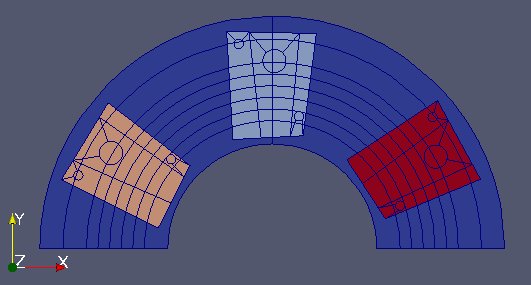
MEDCoupling / MEDLoader - Exemple complet 2 - RJH¶
Ici nous partons de deux fichiers MED très librement inspirés du réacteur expérimental RJH (Réacteur Jules Horowitz).
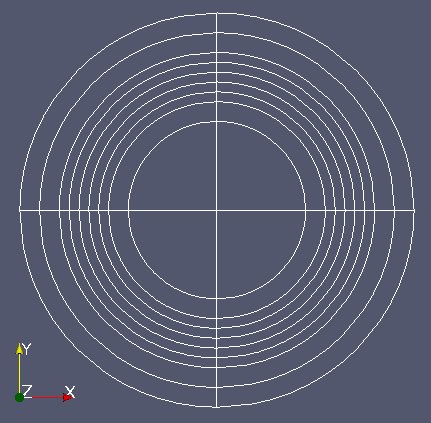
Le premier, Fixe.med, représente la partie statique du réacteur sans dispositif expérimental.
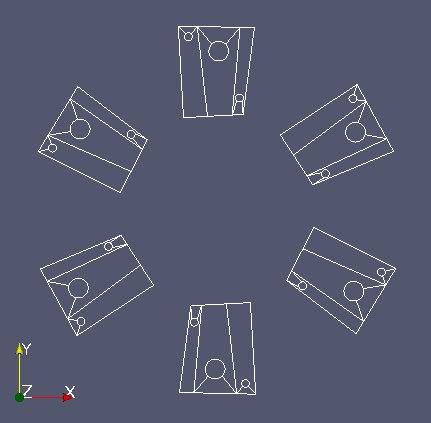
Le deuxième, Mobile.med, représente la partie mobile.
Objectif¶
Le but ici est d’utiliser MEDCoupling pour:
- intersecter ces deux maillages,
- y mettre un champ,
- et ainsi localiser les zones de recouvrement
Début de l’implémentation¶
Pour commencer l’exercice importer tout le module python MEDLoader (qui inclus MEDCoupling).
import MEDLoader as ml
Lire et réparer le maillage statique “Fixe.med”¶
Avec l’API avancée lire tout le fichier “Fixe.med” et appeler fixm l’objet de type MEDCouplingUMesh représentant le maillage statique.
fixe = ml.MEDFileMesh.New("Fixe.med")
fixm = fixe.getMeshAtLevel(0)
Pour ce qui suit il faut absolument que deux cellules se touchant partagent les mêmes edges. Pour ce faire, comme on est en connectivité nodale, il faut absolument que les noeuds soient les mêmes. Il s’avère que cela n’est pas le cas ici. Fusionner le noeuds distants de moins de 1e-10 et regarder l’impact sur le nombre de noeuds de fixm.
print "Nb of nodes in the file : %i " % (fixm.getNumberOfNodes())
fixm.mergeNodes(1e-10)
print "Nb of non duplicated nodes : %i" % (fixm.getNumberOfNodes())
Même traitement pour Mobile.med, le lire avec l’API avancée de MEDLoader (appeler mobm l’instance du maillage) et le réparer en supprimant les noeuds dupliqués.
mobile = ml.MEDFileMesh.New("Mobile.med")
mobm = mobile.getMeshAtLevel(0)
mobm.mergeNodes(1e-10)
Le maillage du RJH étant plus général que des TRI6 et des QUAD8, on a besoin de stocker ces cellules avec un type géométrique à QPOLYG (Quadratic Polygon) qui représente un polygone quadratique (le terme n’est pas très heureux, encore des raisons historiques, ...), c’est-à-dire un polygone avec un nombre arbitraire de côtés, et potentiellement des côtés en forme d’arcs de cercles plutôt que de segments de droites. Ce type géométrique NORM_QPOLYG est dans MEDCoupling/MEDLoader et aussi dans MED fichier.
Nous voudrions visualiser ces deux maillages dans PARAVIS/ParaView, mais nous rencontrons ici deux soucis:
les polygones non-convexes sont, par défaut, mal représentés par VTK en mode Surface. Il faut sélectionner l’option avancée “Triangulate” dans le panneau Display de PARAVIS/ParaView pour avoir un rendu correct.
les arcs de cercles ne sont pas correctement supportés par ParaView. Il faut les tesséliser, c’est-à-dire les transformer en plusieurs petits segments de droite. La méthode MEDCouplingUMesh.tessellate2D() fait ce travail, mais modifie le maillage. Nous faisons donc une copie préalable. Nous passons en paramètre la finesse de découpage (0.1 est suffisant – angle en radian). Attention donc à ne pas modifer ni fixm ni mobm !
fixm2 = fixm.deepCopy() # tessellate2D() modifies the current mesh fixm2.tessellate2D(0.1) fixm2.writeVTK("fixm2.vtu") mobm2 = mobm.deepCopy() mobm2.tessellate2D(0.1) mobm2.writeVTK("mobm2.vtu")
Faire une petite méthode displayVTK(), faisant le travail qui nous servira souvent après.
def displayVTK(m,fname):
tmp = m.deepCopy()
tmp.tessellate2D(0.1)
tmp.writeVTK(fname)
return
Faire des réductions et des repérages de zones¶
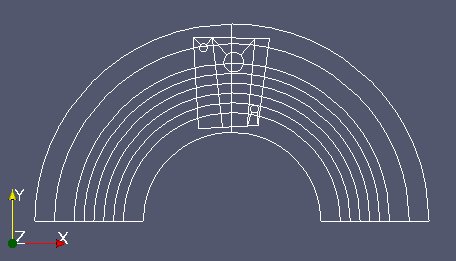
Le maillage mobm est en 6 parties distinctes (voir l’image au dessus). On ne veut récupérer que la première partie. Utiliser MEDCouplingUMesh.partitionBySpreadZone() pour partitionner en zones mobm et ne prendre que la première zone. Appeler cette nouvelle instance zone1Mobm et lui retirer tous les noeuds orphelins (MEDCouplingUMesh.zipCoords()) Enfin l’afficher :
zonesInMobm = mobm.partitionBySpreadZone()
print "Nb of zones in mobm : %i" % (len(zonesInMobm))
zone1Mobm = mobm[zonesInMobm[0]]
zone1Mobm.zipCoords()
displayVTK(zone1Mobm, "zone1Mobm.vtu")
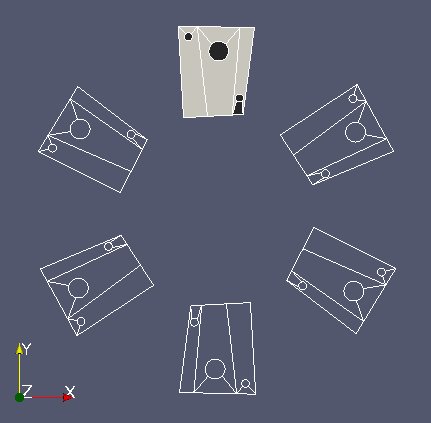
Nous allons désormais travailler autour de zone1Mobm. Nous allons réduire la zone de travail de fixm autour de zone1Mobm. Pour ce faire, réduire fixm en ne prenant que les cellules dans la boîte englobante de zone1Mobm (MEDCouplingUMesh.getBoundingBox() et MEDCouplingUMesh.getCellsInBoundingBox()). Appeler ce nouvel objet partFixm, lui retirer ses noeuds orphelins et l’afficher.
ids2 = fixm.getCellsInBoundingBox(zone1Mobm.getBoundingBox(),1e-10)
partFixm = fixm[ids2]
partFixm.zipCoords()
displayVTK(partFixm,"partFixm.vtu")
Intersecter géométriquement deux maillages¶
C’est le coeur de l’exercice. Nous allons intersecter géométriquement partFixm et zone1Mobm. Cela revient à partitionner à minima partFixm en cellules appartenant soit complètement à partFixm soit à partFixm et zone1Mobm. Invoquer la méthode statique MEDCouplingUMesh.Intersect2DMeshes(), avec partFixm et zone1Mobm et mettre une précision de 1e-10 (seuil de détection de fusion). Cette méthode retourne 3 paramètres (voir API dans la doc) que l’on appellera ici partFixMob, iPart et iMob dans cet ordre.
Sur partFixMob merger les noeuds à 1e-10 près.
partFixMob, iPart, iMob = ml.MEDCouplingUMesh.Intersect2DMeshes(partFixm,zone1Mobm,1e-10)
partFixMob.mergeNodes(1e-10)
Récupérer et afficher la partie de partFixm qui n’est pas dans zone1Mobm. Appeler ce maillage partFixmWithoutZone1Mobm.
ids3 = iMob.findIdsEqual(-1)
partFixmWithoutZone1Mobm = partFixMob[ids3]
displayVTK(partFixmWithoutZone1Mobm,"partFixmWithoutZone1Mobm.vtu")
Maintenant, on va vérifier la qualité du résultat retourné par MEDCouplingUMesh.Intersect2DMeshes(). Pour ce faire on va passer 3 tests:
- Check #0 la somme des aires des cellules de partFixm et égale à celle de partFixMob
- Check #1 la somme des aires des cellules de zone1Mobm et égale à la somme des cells de partFixMob dont l’id dans iMob est different de -1
- Check #2 pour chaque cellule de partFixm, son aire est égale à la somme des aires des cellules de partFixMob
L’aire est une valeur algébrique. Donc attention cette verification ne peut se faire que si les cellules sont toutes bien orientées ou à minima toutes orientées de la même manière. Pour ce faire, regardons les aires des 38 cellules de partFixm (nom de variable : areaPartFixm).
areaPartFixm = partFixm.getMeasureField(ml.ON_CELLS).getArray()
print areaPartFixm.getValues()
On voit que toutes les valeurs sont négatives. Bilan: ce fichier MED ne respecte pas la convention MED fichier ! partFixm étant mal orienté, et MEDCouplingUMesh.Intersect2DMeshes() conservant l’orientation, partFixMob est lui aussi mal orienté. Bref, on va faire les comparaisons sur des tableaux de valeurs absolues. Vérifier alors Check #0.
areaPartFixm = partFixm.getMeasureField(ml.ON_CELLS).getArray()
areaPartFixm.abs()
areaPartFixMob = partFixMob.getMeasureField(ml.ON_CELLS).getArray()
areaPartFixMob.abs()
val1=areaPartFixm.accumulate()[0]
val2=areaPartFixMob.accumulate()[0]
print "Check #0 %lf == %lf with precision 1e-8? %s" % (val1,val2,str(abs(val1-val2)<1e-8))
On peut passer au Check #1. L’esprit est le même que le Check #0.
areaZone1Mobm = zone1Mobm.getMeasureField(ml.ON_CELLS).getArray()
areaZone1Mobm.abs()
val3 = areaZone1Mobm.accumulate()[0]
ids4 = iMob.findIdsNotEqual(-1)
areaPartFixMob2 = areaPartFixMob[ids4]
val4 = areaPartFixMob2.accumulate()[0]
print "Check #1 %lf == %lf with precision 1e-8 ? %s" % (val3,val4,str(abs(val3-val4)<1e-8))
Puis le Check #2.
isCheck2OK = True
for icell in xrange(partFixm.getNumberOfCells()):
ids5 = iPart.findIdsEqual(icell)
areaOfCells = areaPartFixMob[ids5]
areaOfCells.abs()
if abs(areaOfCells.accumulate()[0] - areaPartFixm[icell]) > 1e-9:
isCheck2OK = False
pass
pass
print "Check #2? %s" % (str(isCheck2OK))
Utiliser les informations de l’intersection pour en faire des champs¶
OK pour partFixMob. Nous souhaitons maintenant créer un champ représentant une fonction indicatrice de la zone
Maintenant créer un champ aux cellules sur partFixMob en mettant 0 sur la partie exclusive partFixm et 1 sur la partie couverte. Nous créons donc un champ représentant une fonction indicatrice. Le visualiser en utilisant un fichier VTK (ne pas oublier l’option Triangulate de ParaView).
f = ml.MEDCouplingFieldDouble(ml.ON_CELLS,ml.ONE_TIME)
m = partFixMob.deepCopy()
m.tessellate2D(0.1)
f.setMesh(m)
arr = ml.DataArrayDouble(partFixMob.getNumberOfCells(),1)
arr[iMob.findIdsEqual(-1)] = 0.
arr[iMob.findIdsNotEqual(-1)] = 1.
f.setArray(arr)
f.checkConsistencyLight()
f.setName("Zone")
ml.MEDCouplingFieldDouble.WriteVTK("Zone.vtu",[f])
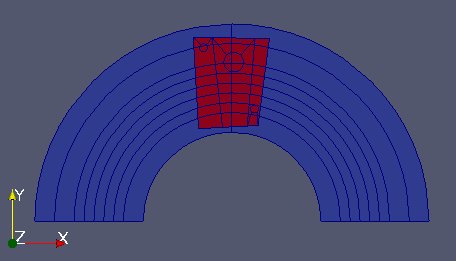
Plus généralement prendre les zones 0, 1 et 5. Faire un champ aux cellules qui vaut 0 dans la zone exclusivement de fixm, 1 dans zone #0, 2 dans la zone #1 et finalement 3 dans la zone #5.
zonesMobm = ml.MEDCouplingUMesh.MergeUMeshesOnSameCoords([mobm[zonesInMobm[0]], mobm[zonesInMobm[1]], mobm[zonesInMobm[5]]])
zonesMobm.zipCoords()
partFixMob2,iPart2,iMob2 = ml.MEDCouplingUMesh.Intersect2DMeshes(partFixm,zonesMobm,1e-10)
partFixMob2.mergeNodes(1e-10)
f2 = ml.MEDCouplingFieldDouble(ml.ON_CELLS, ml.ONE_TIME)
m2 = partFixMob2.deepCopy()
m2.tessellate2D(0.1)
f2.setMesh(m2)
arr = ml.DataArrayDouble(partFixMob2.getNumberOfCells(),1)
arr[iMob2.findIdsEqual(-1)]=0.
st = 0
end = st + len(zonesInMobm[0])
arr[iMob2.findIdsInRange(st,end)] = 1.
st += len(zonesInMobm[0]) ;
end = st + len(zonesInMobm[1])
arr[iMob2.findIdsInRange(st,end)] = 2.
st += len(zonesInMobm[1])
end = st + len(zonesInMobm[2])
arr[iMob2.findIdsInRange(st,end)] = 3.
f2.setArray(arr)
f2.checkConsistencyLight()
f2.setName("Zone2")
ml.MEDCouplingFieldDouble.WriteVTK("Zone2.vtu",[f2])
Ne pas oublier l’option Triangulate de ParaView dans le panneau Display pour bien voir les champs: