Note de travail concernant l’utilisation de MEDMEM¶
Le module MED de SALOME comporte plusieurs composants d’intérêt pour la manipulation de champs:
- la bibliothèque MEDMEM qui fournit une interface de programmation pour manoeuvrer une structure MED
- le module CORBA SALOME_MED qui matérialise le composant SALOME (serveur corba) du module MED
- l’interopérabilité avec le module VISU qui permet la visualisation des champs manipulés dans MED
Les sections ci-après donnent quelques éclairages techniques sur ces différents aspects. Les sources de démonstration peuvent être récupérés depuis le dépôt svn:
$ svn export svn://nepal.der.edf.fr/OM/manifield/trunk manifield
$ svn export svn://nepal.der.edf.fr/FIELD/demofield/trunk demofield
Sommaire
Présentation synthétique de MED¶
MED désigne un modèle conceptuel pour décrire des données de type éléments finis (éléments finis, volumes finis et éléments discrets). Dans l’usage courant, il permet la description et l’échange des données de calcul de type maillages et champs. La documentation complète peut être trouvée à l’URL suivantes:
- Modèle MED (version 2.3).
On distingue deux implémentations informatiques de ce modèle:
- MED fichier: qui permet la lecture et l’écriture de données depuis un fichier au format med. Les opérations de lecture/écriture sont atomiques (pas de chargement de la structure de données globale).
- MED mémoire (noté MEDMEM): qui permet le chargement en mémoire d’une image de la structure de données MED contenue dans un fichier au format med. Les opérations peuvent être atomiques ou globales.
On notera simplement ici que MEDMEM utilise MED fichier pour les opérations de lecture/écriture et que MED fichier est indépendant de MED mémoire. La documentation complète de MED fichier peut être trouvée à l’URL suivante:
La bibliothèque MEDMEM¶
Le modèle de classes MEDMEM est structuré autour des notions de MESH (les maillages), de SUPPORT (le profil des entités) et de FIELD (les champs). Ces notions reprennent en partie des concepts du modèle MED. Le diagramme ci-dessous présente les classes principales:
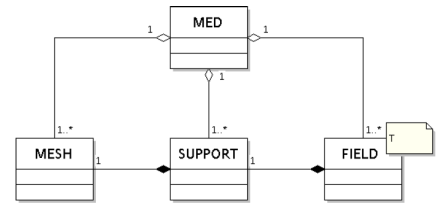
Le conteneur de plus haut niveau dans MEDMEM est la classe MED. La figure ci-dessous indique qu’une instance MED peut être associée à plusieurs maillage et plusieurs champs. Par contre un champ donné ne peut être associé qu’à un seul maillage (par l’intermédiaire du support). Plusieurs champs peuvent être associés au même maillage. La forme la plus courante est d’ailleurs une instance composé d’un maillage unique sur lequel sont définis plusieurs champs.
On peut avoir également des configurations plus complexes, comme par exemple un maillage unique, plusieurs champs définis sur ce maillage mais avec des supports différents, par exemple parce que les valeurs sont définies sur des entités de maillage différentes (les éléments pour un champ, les noeuds pour un autre, ...):
field1->support1->mesh
field2->support2->mesh
field3->support3->mesh
On observe:
- 2 champs U et V doivent avoir le même support (au sens informatique du terme) pour pouvoir être en argument d’une opération (sinon exception). Il faudrait accepter qu’il soit informatiquement différent et vérifier la conformité conceptuelle.
- Cette contrainte peut se comprendre car physiquement les données sont stockées dans un vecteur qui couvre toutes les mailles. Le support est le seul masque de lecture pour établir la correspondance avec les positions dans le maillage et il est donc important qu’une cohérence soit assurée.
Les objets champs (FIELD) et maillage (MESH)¶
Un objet MED permet d’accéder aux différentes informations concernant les objets MESH, SUPPORT et FIELD, mais il ne permet pas d’accéder aux données physiques associées à ces objets (les valeurs des composantes pour les champs, les mailles et leur connectivité pour les maillages). L’accès aux données physiques est du ressort des objets spécifiques MESH, SUPPORT et FIELD.
Un objet MED peut être créé intégralement en mémoire. L’usage plus fréquent est de l’initialiser à partir de la donnée d’un fichier med. Pour cela, l’objet MED doit être associé à un driver d’entrée/sortie branché sur le fichier (testfilename dans l’exemple):
MED *myMed = new MED;
MED_MED_RDONLY_DRIVER *driverIn = new MED_MED_RDONLY_DRIVER(testfilename, myMed);
driverIn->open();
driverIn->readFileStruct();
driverIn->close();
A l’occasion de la fonction readFileStruct, la structure interne de l’objet MED est enrichie des informations concernant les objets MESH, SUPPORT et FIELD contenu dans le fichier. En particulier un dictionnaire des champs (variable map interne) est initialisé est contient l’ensemble des objets FIELD_ préchargés (i.e. avec les méta-données uniquement). Chaque objet FIELD_ ainsi préchargé est autonome pour être chargé sur demande. On peut alors requêter l’objet MED pour obtenir un champ particulier (spécifié par son nom fieldname dans l’exemple):
FIELD<double> *field = (FIELD<double> *)myMed->getField(fieldname, dt, it);
Puis le champ qui lui est associé doit être physiquement chargé pour permettre la mise à jour du support:
MESH * mesh = myMed->getMesh(field);
mesh->read();
myMed->updateSupport();
Pour enfin charger les valeurs des composantes du champ:
field->read();
La numérotation des éléments de maillage¶
Les éléments qui composent un maillage sont caractérisés par:
- Le type d’entité de l’élément, à choisir dans la liste MED_EN::medEntityMesh, qui contient en particulier MED_NODE, MED_FACE, MED_CELL.
- Le type de géométrie de l’élément, à choisir dans la liste MED_EN::medGeometryElement, qui contient en particulier MED_NONE, MED_TRIA3, ..., MED_ALL_ELEMENTS.
Les éléments sont numérotés par un indice relatif à la catégorie géométrique à laquelle ils appartiennent. Ainsi, si le modèle est composé de Na arrêtes et Nf faces de type géométrique MED_QUAD4, alors ces faces sont numérotées de 1 à Nf dans le modèle MED (et de manière persistente dans le fichier med). De même, les arrêtes sont numérotées de 1 à Na. Une numérotion globale implicite existe sur les éléments, elle consiste à parcourir l’ensemble des types géométriques dans l’ordre de définition du modèle de données. Ainsi, si le modèle contient uniquement les Na arrêtes et les Nf faces, alors l’indice global de la première face est Na+1.
Note
Des exemples de code sont disponibles dans le package demofield, fichier python/pybasicfields/MEDMEM_tester.py.
Binding python de MEDMEM¶
Les classes du package MEDMEM (package du module MED qui implémentent les structures de données C++ de MED mémoire) produisent la bibliothèque libmedmem.so. Cette ensemble de classes est en partie mis à disposition de l’interface python grace à une couche de liaison (binding Python-C++) générée par le logiciel SWIG à partir d’un fichier de description d’interface libMEDMEM_Swig.i (dans le package source MEDMEM_SWIG).
Ce fichier d’interface doit être mis à jour dés lors qu’une évolution des interfaces publiques des classes C++ MEDMEM est faite ou qu’une nouvelle classe est créée (du moins si l’on souhaite profiter de ces évolutions dans l’interface python).
Cette mise à jour nécessite de prendre soin au transfert des structures de données entre les espaces python et C++. En particulier, l’utilisation des template de classe pour décrire les champs typés en C++ appelle une précaution de codage particulière de l’interface SWIG.
Pour exemple, le fragment de code ci-dessous, extrait du fichier libMEDMEM_Swig.i, montre comment déclarer la nouvelle classe MedDataManager dans l’interface:
#include "MEDMEM_MedDataManager.hxx"
class MedDataManager
{
public:
~MedDataManager();
void printFieldDouble(FIELD<double,FullInterlace> * field);
%extend {
MedDataManager(char * fileName)
{
return new MedDataManager(string(fileName));
}
MedDataManager(MED * med)
{
return new MedDataManager(med);
}
%newobject getFieldDouble(const char * fieldName, const int dt, const int it);
FIELD<double, FullInterlace> * getFieldDouble(const char * fieldName, const int dt, const int it)
{
return (FIELD<double, FullInterlace> *) self->getFieldDouble(string(fieldName), dt, it);
}
}
};
Utilisation de MEDMEM pour la manipulation de champs¶
Des opérations de manipulation de champs sont disponibles dans la bibliothèque MEDMEM standard est peuvent être utilisées dans l’interface python. Les quelques lignes suivantes illustrent l’usage qu’on peut en faire pour exécuter l’addition de deux champs sur tout leur espace de définition et pour un pas de temps donné:
from libMEDMEM_Swig import MedDataManager
from xmed.helper import readMed, writeMed
# Load the medmem data structure from a med file
med = readMed("/tmp/input.med")
# Then create a med data manager to deal with the fields data
dm = MedDataManager(med)
# Get the timestamps (dt,it)=(-1,-1) of the fields "testfield1" and "testfield2"
f1 = dm.getFieldDouble("testfield1",-1,-1)
f2 = dm.getFieldDouble("testfield2",-1,-1)
# Create a new field as the sum of f1 and f2
r = f1 + f2
# And add this new field to the med data structure
med.addField(r)
# Finally, write the whole data in an output med file
writeMed(med,"/tmp/output.med")
Note
Cet exemple de code requiert les évolutions de MEDMEM opérées dans la branche BR_medop (pour disposer de la classe MedDataManager en particulier) et le package python xmed qui fournit quelques fonctions utilitaires pour manoeuvrer les données med (ce package est dans le module XMED et sera probablement à terme intégré au module MED).
Des limitations existent aujourd’hui pour ce type de manipulations:
- les champs doivent partager le même support MED, c’est-à-dire être décrit sur le même maillage et sur les mêmes entités de ce maillage.
- ...
Remarque sur l’implémentation C++¶
A noter l’usage de plusieurs formes d’arguments pour les fonctions:
- passage des arguments par valeur myfunction(A a);
- passage des arguments par référence myfunction(A& a);
- passage des arguments par pointeur myfunction(A* a);
Le passage des arguments par référence est une facilité d’écriture pour éviter de passer un pointeur tout en évitant la récopie des données de la variable.
L’interface CORBA SALOME_MED¶
Implémentation du composant MED et des servants SALOME_MED::*¶
Le composant MED est un servant CORBA qui permet la manipulation de données MEDMEM dans l’environnement SALOME. Le composant peut fournir des pointeurs vers des instances de l’interface SALOME_MED (objets SALOMEMED::MED, SALOME_MED_FIELD, ...). Ces instances sont des servants CORBA qui résident dans le container et qui encapsulent les données MEDMEM.
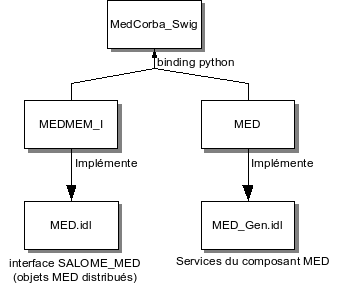
Le schéma ci-dessous représente les éléments informatiques qui composent l’architecture CORBA du module MED:
Les structures MEDMEM (données physiques) et SALOME_MED (wrapping CORBA) fonctionnent différement en ce qui concerne le chargement des données:
- Dans MEDMEM, les données sont chargées à la demande (fonctions read des objets) et aucune gestion n’est assurée. En particulier l’appel à read alors que la donnée est déjà chargée conduit à une levée d’exception. C’est à l’utilisateur de MEDMEM de prendre en charge ce type de gestion.
- Dans SALOME_MED, les données sont chargées à la création de l’instance SALOME_MED::MED. Les maillages ainsi que les champs et leurs données sont chargés à ce moment là et gérés dans une table de type HashMap au niveau de la structure SALOME_MED::MED. Cette structure remplie dés lors des fonction de gestion. L’appel à SALOME_MED::MED.getField(...) ne charge pas les données mais renvoie un pointeur SALOME_MED::FIELD_ptr sur les données chargées à l’initialisation (ATTENTION, cette fonction est bugguée dans la branche principale -> Fix dans la branche BR_medop).
Une gestion intermédiaire peut être envisagée: le chargement à la demande géré dans une ou plusieurs tables de champs (une pour chaque type de valeur numérique). Une implémentation de ce type de gestion est illustré dans la classe MedDataManager du package MEDMEM qui prend en charge ce comportement pour les structures de données MED (en particulier les champs).
Utilisation du composant MED¶
Le module SALOME MED fournit un module CORBA appelé SALOME_MED. Les interfaces de ce module CORBA sont spécifiées par les fichiers idl suivants:
- le fichier [http://nepal.der.edf.fr/pub/SALOME_userguide/MED5/doc/salome/tui/MED/MED_8idl.html MED.idl] qui décrit les interfaces des objets manipulés par le module SALOME_MED. On trouve en particulier les objets MESH, SUPPORT et FIELD.
- le fichier
[http://nepal.der.edf.fr/pub/SALOME_userguide/MED5/doc/salome/tui/MED/MED__Gen_8idl.html
MED_Gen.idl] qui décrit les interfaces du composant SALOME
(c’est-à-dire le composant chargé par la commande
FindOrLoadComponent("FactoryServer", "MED") du
lyfeCycleCorba). On trouve:
- l’interface MED_Gen_Driver qui hérite de SALOMEDS::Driver pour l’implémentation des services généraux des composants SALOME (persistance hdf, dump)
- l’interface MED_Gen qui hérite des interfaces Engines::Component et MED_Gen_Driver pour l’implémentation des services spécifiques du composant MED.
L’implémentation de ces interfaces est faites au niveau de différents packages des sources du module MED:
- Le package MEDMEM_I qui fournit l’implémentation C++ des interfaces décrites par le fichier MED.idl;
- Le package MED qui fournit l’implémentation C++ des interfaces décrites par le fichier MED_Gen.idl, et qui correspond à la partie composant classique d’un module SALOME.
- Le package MedCorba_Swig qui fournit une interface swig générée à partir de l’implémentation C++ de MEDMEM_I et MED
L’utilisation peut être illustrée au moyen d’exemples python (i.e. qui utilise l’interface swig fournie par MedCorba_Swig). Après l’import d’amorce systématique:
import salome
salome.salome_init()
import SALOME_MED
from libSALOME_Swig import *
On peut charger le composant SALOME MED:
medComp=salome.lcc.FindOrLoadComponent("FactoryServer", "MED")
grâce auquel les services de chargement de la structure MED peuvent être invoqués. Par exemple, les commandes suivantes chargent toute la structure MED dans l’étude salome passée en argument:
filePathName = "myfile.med"
medComp.readStructFileWithFieldType(filePathName,salome.myStudyName)
Ce deuxième exemple charge la structure MED mais ne place pas le résultat dans l’étude:
filePathName = "myfile.med"
medObj = medComp.readStructFile(filePathName,salome.myStudyName)
On récupère à la place un objet de classe SALOME_MED::MED qui permet une utilisation assez semblable (mais différente on le verra plus bas) à MEDMEM:
fieldIdx = 1 # WRN maybe there is no field of idx=1
iterationIdx = 0
fieldName = medObj.getFieldNames()[fieldIdx]
dtitfield = medObj.getFieldIteration(fieldName,iterationIdx)
it = dtitfield[0]
dt = dtitfield[1]
fieldObj = medObj.getField(fieldName,it,dt)
nbOfFields = medObj.getNumberOfFields()
fieldNames = medObj.getFieldNames()
mesh = fieldObj.getSupport().getMesh()
Note
Observations en vrac:
- Un FIELD_i possède un champ de type MEDMEM::FIELD_ qui représente le champ informatique réel (objet MEDMEM).
- FIELD_i::fieldMap variable static de type map qui semble gérer les différentes instances de FIELD_i (~pattern factory). Cette map peut être requétée au moyen d’un indice de type long appelé corbaIndex.
- Quand on crée un FIELD_i par le constructeur d’argument MEDMEM::FIELD_, le MEDMEM::FIELD_ est ajouté dans la map avec incrément du corbaIndex
- La fonction FIELD_i::read(i) redirige vers la fonction read(i) du MEDMEM::FIELD_ associé
- A CONFIRMER: Il semble que les fonctions de chargement readStructFile*() charge toutes les données du fichier med, alors qu’en MEDMEM seules les meta-données sont chargées.
- A CONFIRMER: il semble que le chargement d’une structure MED CORBA peut se faire sans passer par le composant (cf. l’interface de MED)
Interface avec le module VISU¶
Des interactions sont possibles entre MED et VISU à partir du moment où les données med sont gérées dans l’étude, c’est-à-dire sous la forme d’objets SALOME_MED (voir ci-dessus) publiés dans l’étude. Les deux conditions sont aujourd’hui nécessaires (objet corba + publié dans l’étude) mais il semble que ce ne soit lié qu’à un choix d’interface VISU (la fonction ImportMed en particulier) qui peut a priori être modifié. A CONFIRMER.
L’exemple de code ci-dessous (en python, mais il peut être transposé à une implémentation C++) montre par exemple comment envoyer au module VISU une requête de visualisation d’un champs hébergé par le module MED (en fait, les données sont gérées au travers d’un objet corba SALOME_MED “délocalisé” et qui a été référencé dans l’étude dans la catégorie du composant MED). Les importations standard (salome, SALOME_MED, ...) sont supposées avoir été faites au préalable (voir les exemples précédents):
# Load the med structure using MED
medComp=salome.lcc.FindOrLoadComponent("FactoryServer", "MED")
filePathName = "myfile.med"
medComp.readStructFileWithFieldType(filePathName,salome.myStudyName)
# Get the VISU component
import VISU
visuComp = salome.lcc.FindOrLoadComponent("FactoryServer", "VISU")
visuComp.SetCurrentStudy(salome.myStudy)
# Get the sobject associated to the med object named "Med"
aSObject = salome.myStudy.FindObject("Med")
isPresent, medSObj = aSObject.FindSubObject(1)
# Finally, import the med sobject in VISU
result = visuComp.ImportMed(medSObj)
Il est possible de d’aller plus loin et par exemple de déclencher l’affichage d’une scalarmap d’un champ spécifique pour une itération particulière (voir la fonction TEST_SALOMEMED_requestToVisu_scalarmap du fichier SALOMEMED_tester.py fourni dans les sources d’exemple).
Liens complémentaires:
- http://nepal.der.edf.fr/pub/SALOME_userguide/VISU_V5_1_3/doc/salome/gui/VISU La documentation utilisateur en ligne du module VISU
Notes en vrac¶
Questions:
- Comment obtenir le nom du fichier med à partir d’une structure med?
- Peut-on imaginer un moyen de fournir l’objet MEDMEM::MED à partir de la donnée de l’objet CORBA SALOME_MED::MED?
Remarques:
- A part, les opérations arithmétiques (+,-,*,/), aucune opération n’est définie.